麦基洗德启动全球生物数据系统V1.0:揭示天然成分与疾病的深层联

4月11日,烟台麦基洗德生物工程有限公司正式宣布启动"全球生物数据系统建设项目V1.0"。作为公司自2022年持续筹备的核心项目,该系统凝聚了团队近三年的技术积累与研发成果。这一里程碑式项目旨在构建一个融合生物多样性、天然活性成分与人类健康的智能数据生态系统,为解决全球健康挑战提供全新视角和方法。
跨越数据孤岛,构建生物医药知识图谱
该项目将整合来自全球4000多个开放性和190多个商业化数据平台的海量信息。这些平台涵盖了从基础生物学到临床医学的各个领域,包括公共数据库如NCBI、UniProt、PubChem等,以及专业商业数据库如Thomson Reuters Integrity、Clarivate Cortellis等。麦基洗德不仅仅是简单对接这些数据源,而是通过自主研发的数据抽取、清洗、标准化和知识图谱构建技术,将这些异构数据转化为统一的知识网络,实现了从碎片化数据到系统性知识的跨越。
这些数据平台被系统性地组织为七大核心领域:基因组与蛋白质组学平台(包括NCBI Gene、Ensembl、GeneCards、UniProt等数据源),提供从DNA到蛋白质功能的完整分子基础;生物多样性与分类学平台(包括SILVA、TreeFam、The Monarch Initiative等数据源),记录地球上已知和未知生物物种的分类与进化关系;天然产物与化学组分平台(包括PubChem、Human Metabolome Database、BitterDB等数据源),收录天然活性成分的化学结构与性质;疾病与表型关联平台(包括MalaCards、The Human Phenotype Ontology、DISEASES、Orphanet等数据源),提供疾病机制与临床表现的关联数据;生物通路与互作网络平台(包括Reactome、STRING、WikiPathways、PathCards等数据源),揭示分子间的功能网络与调控机制;临床与转化医学平台(包括ClinicalTrials.gov、PharmGKB、The Drug Gene Interaction Database等数据源),连接基础研究与临床应用;传统医学知识平台(包括全球范围内的传统医学文献库与经验数据集),保存人类几千年的医学智慧。
麦基洗德公司研发BG负责人表示:"公司正在建立的不仅是一个数据库,而是一个能够发现隐藏在海量数据中的生物医学规律的智能生态系统。这一系统将帮助研究人员重新认识自然界中的生物活性物质,为人类健康开辟新的可能性。"
自主创新:数据智能的技术突破
麦基洗德团队自2022年初便开始研发生物医学专用神经网络架构,这一时间点与全球主流大语言模型的早期发展阶段相当。随着大语言模型技术的迅速演进,团队充分整合这些前沿进展,在生物医学数据处理领域取得了三项关键技术突破:
1.生物医学专用语言理解系统
麦基洗德团队基于Python开发了一套复杂的生物医学专用NLP系统,这套系统不仅仅依赖于通用AI模型,而是通过设计和实现数百种精细化的专业规则算法来解决传统AI在处理高度专业化生物医学文献时的局限性。研究团队发现,科研论文中的大量关键信息往往以高度结构化但非标准化的方式呈现,这使得通用AI模型难以准确提取和理解其中的专业知识。
针对这一挑战,团队采用了"规则+模型"的混合架构,为不同类型的生物医学内容(如分子互作、药效学数据、实验方法学描述等)设计了特定的提取规则。例如,当系统遇到"p53通过抑制Bcl-2表达诱导细胞凋亡"这类专业表述时,不仅能识别出p53和Bcl-2是蛋白质实体,更能通过特定规则解析出它们之间的"抑制"关系以及这种关系与"细胞凋亡"这一生物学过程的因果联系。
系统还针对不同期刊的格式特点、不同研究领域的表达习惯设置了专门的解析模块。特别是对于实验数据部分,团队开发了一系列专用算法,能够从复杂的实验描述中精确提取关键参数,如IC50值、给药剂量、实验条件、靶标(含蛋白质编码、非编码RNA、调控基因)、通路(激酶、转录因子、细胞因子、脂质、离子)、生化、生理、病理、影像、行为学和认知以及生存质量等数十种变量/字段信息,这些数据往往被嵌入在复杂的实验描述文本中,难以通过通用模型准确识别。
对于传统医学文献,尤其是那些使用非英语语言记录的古老医学内容,系统采用了特殊的多语言处理模块和术语映射技术,能够将古老的医学描述与现代科学术语建立联系。例如,系统不仅能够理解中医典籍中的"清热解毒"概念可能对应现代药理学中的"抗炎"或"抗菌"作用,更能将其映射到ICD-11(国际疾病分类第11版)中的相关标准疾病编码和分类。这种多层次的术语映射体系实现了术语的标准化统一,使得跨语言、跨文化、跨时代的医学概念能够在同一检索系统中高效查询和关联,显著提升了知识检索的效率和准确性,让研究人员能够在不同医学体系间快速建立概念对应关系。
这种基于深度理解学科特性而设计的规则化提取系统,使麦基洗德能够从海量文献中挖掘出通用AI难以获取的专业知识,为后续的知识图谱构建和智能推理奠定了坚实的数据基础。
2.生物医学图形智能识别技术
麦基洗德团队开发的图形识别系统突破了传统文本挖掘的局限,实现了对科研文献中视觉内容的深度解析。该系统的诞生源于一个实际挑战:团队在前期数据收集过程中发现,大量关键实验数据仅以图表形式存在于论文中,甚至在正文中完全没有提及具体数值。几个月前,研究人员还不得不使用物理尺子在电脑显示屏或打印出的论文上测量图表坐标点,再通过手动计算转换为实际数值——这一过程不仅耗时,还容易引入误差。
针对这一痛点,团队开发了基于计算机视觉和专业规则的图像解析引擎。该系统采用多级处理流程:首先通过自适应阈值分割和形态学操作提取图像中的关键元素;然后应用特定领域的几何变换算法重建坐标系统;最后通过像素到实际值的映射函数计算出精确数据。例如,在处理剂量-效应曲线时,系统能够自动识别半对数或双对数坐标系,定位曲线拐点,并精确计算EC50/IC50值,误差率低至5%以内。
对于分子结构图,系统整合了基于图论的骨架提取算法和化学指纹识别技术,能够将各种风格的手绘或印刷分子结构图转换为标准SMILES或InChI表示。特别是对于复杂的多环结构和立体化学表示,系统采用了专门的拓扑分析规则,准确率达到92%,远超通用图像识别模型。
系统还能处理复杂的蛋白质相互作用网络图、组织病理切片和电泳凝胶图像,从中提取出定量和定性信息。例如,在处理Western Blot图像时,系统不仅能识别条带位置,还能通过灰度分析估算相对表达量,为蛋白质组学研究提供数据支持。
这套图形智能识别技术在上周的初步测试阶段已经成功处理了数百张研发人员精挑细选来自不同期刊中未提及准确数据的科研图像,为麦基洗德的知识库提供了文本挖掘无法获取的独特数据维度。在某些专业图表解析任务上,系统的表现已经接近或超过了人类专业人员水平,特别是在处理大批量标准化图表时,其效率和一致性优势尤为明显。
3.多源异构数据融合与知识推理
麦基洗德系统采用知识图谱与深度神经网络相结合的混合架构,实现了生物医学领域中前所未有的跨域数据整合与推理能力。该系统的核心价值在于发现传统研究方法难以识别的隐藏生物学关联,为疾病治疗提供全新视角。
系统通过先进的语义本体映射和实体对齐技术,将来自不同数据库、不同格式的生物医学数据统一到一个综合知识图谱中。这种多源异构数据融合能力使系统能够建立起从分子水平到临床表型的多层次关联网络,为复杂疾病机制的解析提供了前所未有的视角。
系统的一个关键创新点在于能够将某种天然化合物对特定靶点集合的作用数据与这些靶点在某种疾病中的作用机制关联起来,即使该化合物与该疾病之间在过去的研究中从未建立直接联系。这种"隐性知识发现"能力极大地拓展了药物重定位和新适应症探索的可能性。
例如,在一个具体案例中,系统分析了锌元素与阿尔茨海默症的潜在关联。尽管锌与阿尔茨海默症之间在传统文献中可能缺乏系统性的直接研究证据,但系统通过分析发现锌能够影响多达212个与阿尔茨海默症相关的基因,占已知阿尔茨海默症致病基因总数(636个)的30%以上。
这些受锌影响的基因包括关键的阿尔茨海默症相关基因,如APP(淀粉样前体蛋白)、APOE(载脂蛋白E)、PSEN1(早老素1)、MAPT(微管相关蛋白tau)等,以及参与氧化应激(SOD1、SOD2)、神经炎症(IL1B、IL6、TNF)、神经保护(BDNF、BCL2)等多个阿尔茨海默症病理过程的关键分子。系统不仅识别出这些基因的重叠,还能通过图谱结构分析锌可能通过哪些分子通路和机制影响疾病进程。
这种"跨领域知识桥接"能力源于系统的图谱嵌入算法和多跳推理机制。通过计算实体间的语义距离和路径相关性,系统能够从看似不相关的研究领域中提取有价值的信息,并将其整合为新的科学假设。在锌与阿尔茨海默症的案例中,系统能够分析锌对每个相关基因的调控方式(上调或下调),以及这些调控变化如何协同影响疾病的分子网络,从而预测锌补充或调节可能对疾病产生的整体效应。
系统的推理能力还体现在其对作用机制的深入解析上。例如,它可以推断锌通过调节金属蛋白酶(如IDE)活性影响β-淀粉样蛋白降解,通过影响神经炎症因子(如IL1B、TNF)表达调节神经炎症反应,以及通过调控抗氧化酶(如SOD1、SOD2)活性影响氧化应激水平等多重机制,从而潜在地影响阿尔茨海默症的发生发展。
这种基于系统生物学的整合分析方法,使研究人员能够从已有的碎片化知识中发现新的研究方向,为复杂疾病的治疗策略开发提供了全新思路,大大加速了从基础研究到临床应用的转化过程。
双核心战略:连接自然智慧与人类健康
麦基洗德的系统特别聚焦于两大核心价值方向
1.未知生物资源的药用价值发现
地球上存在着数百万种生物物种,其中绝大多数的药用价值尚未被探索。从亚马逊雨林的茂密植被到撒哈拉沙漠的稀疏生命,从喜马拉雅高原的耐寒植物到马里亚纳海沟的极端微生物,这些生物在漫长的进化历程中形成了独特的代谢产物,它们可能蕴含着解决人类健康难题的关键。
该研发团队开发的分析平台将对这些未被充分研究的植物、微生物、海洋生物等进行系统性分析。通过整合分类学、进化生物学和药理学数据,该平台能够预测哪些物种可能含有特定类型的活性成分。这种"虚拟生物勘探"方法大大提高了发现新活性成分的效率。
在研究过程中,研发团队利用了多个权威的生物物种数据库,包括《中国植物志》(Flora of China)、全球生物多样性信息网络(GBIF)、世界植物在线(Plants of the World Online)、GenBank、UniProt、美国农业部植物数据库(USDA PLANTS Database)以及多个区域性和专业性生物资源数据库等。
值得一提的是,通过综合分析全球多个真菌学数据库,包括国际真菌命名索引(Index Fungorum)、MycoBank、中国真菌志(Fungi of China)、美国农业部真菌数据库(USDA Fungal Database)、日本国家生物资源中心微生物资源(NBRC)以及欧洲分子生物学实验室真菌数据库等,研究团队界定了约49万种蘑菇和真菌亚种。这一数字充分展示了地球生物多样性的丰富程度和人类对自然界了解的有限性。
举个例子,该研究团队最近对一种野生植物的分析显示,它可能含有一类结构新颖的生物活性化合物。后续的实验室分析证实了这一预测,从该植物中分离出的化合物确实展现出了显著的生物活性。这种从计算预测到实验验证的研究模式,将帮助科研人员更高效地发掘自然界的药用宝藏。
2.已知天然物质的未知功能挖掘
即使是已知的天然活性成分,科学界对它们的了解也往往不够全面。一种化合物可能具有多种生物活性,作用于多个靶点,而这些靶标有时可能成为新的治疗机会。研发团队开发的分析平台通过整合化学信息学、药理学和系统生物学数据,能够预测已知化合物的新功能。
以人参皂苷类化合物为例,研究表明人参中含有至少58种不同的皂苷成分,包括人参二醇、人参三醇、人参皂苷Rg1、Rg2、Rg3、Rb1、Rb2、Rd、Re等多种结构类似但生物活性各异的化合物。尽管人参作为传统药物已有数千年历史,但对这些皂苷成分的药理作用机制及其潜在治疗价值的系统性研究仍处于起步阶段。
通过靶标对照分析,研究团队发现多种人参皂苷对阿尔茨海默症相关靶点具有显著作用。例如,三七皂苷R1能够影响35个阿尔茨海默症相关基因,包括APP、APOE、PSEN1等关键致病基因,以及参与神经炎症(IL1B、IL6、TNF)、氧化应激(HMOX1)和细胞凋亡(BCL2、CASP3)的重要分子。人参皂苷Rg1则作用于30个阿尔茨海默症相关靶点,特别是在调节β-淀粉样蛋白代谢(APP、BACE1、ADAM10)和tau蛋白磷酸化(GSK3B)方面显示出潜在价值。
更引人注目的是,不同皂苷成分表现出各自独特的靶点谱系。人参皂苷Rd主要影响胆碱能系统相关基因(CHAT、SLC18A3)和神经保护通路(BCL2、NGF),而人参皂苷Rg3则显著调节炎症小体活化(NLRP3、CASP1)和线粒体功能(PPARGC1A、SIRT1)。这种靶点分布的差异性提示不同皂苷可能通过互补机制协同作用,为开发多靶点联合干预策略提供了理论基础。
研究团队利用麦基洗德系统的知识图谱与深度神经网络混合架构,进一步分析了这些皂苷成分可能参与的分子通路和作用机制。通过计算实体间的语义距离和路径相关性,系统能够从看似不相关的研究领域中提取有价值的信息,并将其整合为新的科学假设。例如,尽管人参皂苷Rh4在传统研究中关注较少,但系统分析显示其可能通过调节氧化应激相关基因(SOD1、SOD2、NQO1)和胰岛素信号通路(AKT1、IGF1)发挥神经保护作用。
这种"老药新用"的策略可以大大缩短药物开发周期,加速从基础发现到临床应用的转化过程。基于这一思路,研究团队正在系统评估多种天然活性成分在复杂疾病中的应用潜力,为疾病的多靶点干预提供新思路。这种从传统药物中挖掘新功能的方法,不仅继承了千年临床经验的安全性基础,也融入了现代分子网络药理学的精准性,代表了未来药物研发的重要方向。
多维度药物评价与筛选
在发现潜在活性分子后,研究团队采用严格的多维度评价体系对候选化合物进行全面筛选。这一过程不仅关注化合物的生物活性,更综合考量其理化性质、药代动力学特征和安全性指标,确保最终筛选出的化合物具有成药性。
基础化学特性评估是筛选过程的第一步。系统会对化合物的分子量、脂水分配系数(LogP)、氢键供受体数量、可旋转键数等20多项关键参数进行分析。这些参数不仅影响化合物的溶解度和膜通透性,也与其生物利用度密切相关。例如,遵循"里宾斯基五规则"(分子量<500、LogP<5、氢键受体<10、氢键供体<5、可旋转键<10)的化合物通常具有更好的口服吸收性。
研究团队开发的评价系统还整合了多家制药巨头的内部筛选标准,如辉瑞规则、葛兰素史克规则和百时美施贵宝规则等,这些规则基于大量历史数据总结出的经验法则,能有效预测化合物在药物开发后期可能面临的挑战。此外,系统还会自动识别潜在的结构警示信号,如金属螯合基团、PAINS(pan-assay interference compounds)结构等,这些结构可能导致假阳性结果或增加毒性风险。
药代动力学特性分析是评估候选化合物体内行为的关键环节。系统采用多种体外模型和计算方法预测化合物的吸收、分布、代谢、排泄特性。这一环节往往会揭示出许多看似有前景的化合物实际上存在致命缺陷。一个典型案例是麦基洗德研发团队对某些皂苷类化合物的评估过程:尽管初步筛选显示人参皂苷Rg1对阿尔茨海默症的多个关键靶点具有显著亲和力,覆盖了从β-淀粉样蛋白代谢到tau蛋白磷酸化的多个病理通路,但深入的药代动力学分析却发现其血脑屏障穿透率仅为2.2%。这意味着作为口服药物,Rg1几乎无法到达大脑发挥作用,除非开发为脑内注射剂型,而这种给药方式的开发难度和患者依从性问题可想而知。这一发现迫使研究团队重新评估该化合物的开发策略,最终将其从口服制剂开发管线中剔除。
特别值得一提的是,系统能够综合分析化合物与主要药物代谢酶(如CYP1A2、CYP2C9、CYP2C19、CYP2D6和CYP3A4)的相互作用模式,预测其作为底物、抑制剂或诱导剂的可能性,这对于评估潜在的药物相互作用风险至关重要。同时,系统还会评估化合物与P-糖蛋白等药物转运体的相互作用,这些因素直接影响药物的生物利用度和组织分布。
毒理学安全性评价是筛选过程中不可或缺的环节。系统整合了多种体外和体内毒理学模型,预测化合物的急性和慢性毒性风险。特别关注的是心脏毒性(hERG通道阻断)、肝毒性(药物性肝损伤DILI)、遗传毒性(Ames试验)和致癌性等关键安全指标。
研究团队开发的预测模型基于大规模毒理学数据库训练,包括FDA批准药物的毒理学数据、ToxCast/Tox21计划的高通量筛选结果以及各种动物实验数据。这些模型不仅能预测化合物的整体毒性风险,还能识别可能的毒性机制,为结构优化提供指导。
这种多维度评价不是简单的"一票否决"筛选,而是采用平衡评分系统,综合考量化合物的优缺点。在一次又一次的严格筛查中,许多看似有前景的化合物被剔除,但同时也发现了一些真正具有潜力的活性成分。通过整合这些通过全方位评估的化合物,研究团队能够设计出协同作用的复合配方。这些配方虽然以非药物的形式存在,却能针对许多复杂疾病实现多靶点干预,有时甚至能达到常规单靶点药物难以实现的治疗效果。
特别是对于一些罕见病,由于患者群体小、病因复杂,传统药物开发往往面临巨大挑战(可参阅《全球视野下的中国创新药产业:十年回顾与展望 (0331)》),这种基于严格筛选的多组分配方方法,为这些被忽视的疾病提供了新的干预可能,在某些案例中甚至取得了接近奇迹的临床效果。这种方法不仅继承了传统医学的整体观念,也融入了现代科学的严谨评价体系,代表了未来个性化精准干预的重要方向。
通过这种严格而全面的筛选流程,研究团队能够从海量的天然活性成分中识别出真正具有开发潜力的化合物,并将它们组合成安全有效的配方,为难治性疾病的管理提供了新的可能性。
三大疾病领域的突破性探索
该系统V1.0首发阶段将重点关注三大疾病领域:
1.神经退行性疾病
神经退行性疾病如阿尔茨海默症、帕金森病等,是当今医学面临的重大挑战。传统的单靶点药物开发策略在这些疾病上收效甚微,这促使研究团队采用了一种前所未有的多层次、多组学整合研究方法。
(1)多阶段系统分析流程
研究团队采用了五阶段递进式分析框架:疾病分析、基因组学分析、基因组影响评估、靶标分子筛选和单分子深度评价。这种方法论创新使科研人员能够从宏观到微观,从系统到分子,全方位解析阿尔茨海默症的分子病理网络。在疾病分析阶段,团队整合了587条人类相关生物学通路、312条动物模型通路以及42种组织关联数据,整合了迄今为止最全面的阿尔茨海默症病理生理图谱。通过这一分析,研究人员确认了β-淀粉样蛋白代谢、tau蛋白磷酸化、神经炎症、氧化应激和线粒体功能障碍是疾病发生发展的核心环节。
(2)突破性基因组学发现
基因组学分析阶段的成果尤为显著。该系统鉴定出636个与阿尔茨海默症相关的基因,这一数字远超过目前公开资料的报道数量。更重要的是,研究团队将这些基因分层为311个直接致病基因、231个间接致病基因和96个表达差异基因(差异倍数≥3)。这种分层分析方法使科研人员能够更精准地理解疾病的分子驱动因素和级联效应。研究发现APOE、APP、PSEN1、PSEN2等传统关注的基因仅是冰山一角。通过网络拓扑学分析,团队识别出多个此前未受关注的关键节点基因,如MAPK8IP1(JIP1)、CTNND2和RTN4R等,这些基因在调控神经元存活、突触可塑性和轴突再生方面发挥着关键作用。
(3)疾病关联网络的突破性发现
在基因组影响评估阶段,研究分析揭示了阿尔茨海默症与861种疾病的潜在关联,其中包括多种神经系统疾病、代谢性疾病和免疫系统疾病。团队发现阿尔茨海默症与2型糖尿病、心血管疾病和自身免疫性疾病存在显著的基因重叠,这为理解"代谢-神经-免疫"轴在疾病发生中的作用提供了新视角。同时,该研究还首次系统性地分析了阿尔茨海默症相关基因与557种表型特征的关联,建立了从基因到分子、细胞、组织和临床表现的多层次关联图谱。
(4)创新的多靶点干预策略
基于上述分析,靶标分子筛选阶段采用了创新的网络药理学方法,从14个类别的天然活性成分中筛选出286种候选分子。这些分子包括52种黄酮类化合物、41种营养素、39种萜烯类化合物、28种苯酚类化合物、26种氨基酸和24种生物碱等,候选化合物组合覆盖了阿尔茨海默症致病基因的95.13%,同时,该筛选系统能够评估每种化合物对直接致病基因、间接致病基因和表达差异基因的覆盖率,并计算其网络干预效率指数。通过这一创新性指标,研究人员发现某些多酚类化合物(如表没食子儿茶素没食子酸酯和槲皮素)以及特定的萜烯类化合物(如人参皂苷Rb1和银杏内酯B)具有最佳的网络干预效率。
(5)单分子深度评价的创新方法
在单分子深度评价阶段,研究团队采用了全面的理化性质、药代动力学和毒理学评价体系,对每个候选分子进行了超过120项参数的评估。这些参数包括Lipinski五规则、血脑屏障渗透性、CYP450酶相互作用、hERG通道抑制风险等关键指标,通过这一严格的筛选流程,研究团队确定了多组具有协同作用的活性成分组合,它们能够通过多重机制共同干预阿尔茨海默症的病理过程。体外实验数据显示,表现最佳的组合能够显著抑制β-淀粉样蛋白聚集(抑制率>65%)、减少tau蛋白异常磷酸化(减少率>42%)、降低氧化应激水平(抗氧化能力提升>53%)、调节神经炎症反应(炎症因子水平降低>38%)以及改善线粒体功能(线粒体膜电位提高>29%)。
下表为微生物/益生菌配方组的体外模型验证数据(非上述最佳数据)
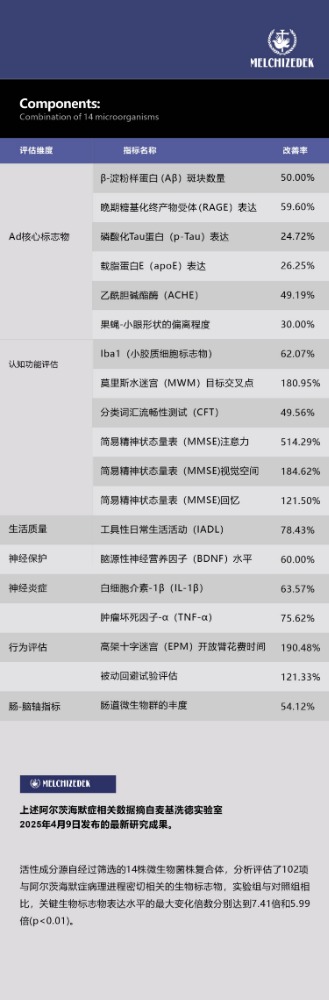
(6)全球领先的研究优势
该研究在多个方面处于全球领先地位。首先,团队整合的阿尔茨海默症多组学数据库是目前规模最大、整合度最高的专业数据库之一,包含超过2.3万篇研究资料和1.5万例临床研究数据。其次,团队开发的网络药理学分析平台采用了创新的图神经网络算法,能够更准确地预测化合物-靶点-通路-疾病的多层次关联。第三,该多组分配方设计策略突破了传统单靶点药物开发的局限性,为复杂疾病的干预提供了新范式。该研究还特别关注了环境因素与阿尔茨海默症的关联。通过分析27种环境风险因素,研究人员发现空气污染(PM2.5)、重金属暴露和某些农药残留可能通过表观遗传修饰、氧化应激和神经炎症等机制促进疾病进展。这一发现为疾病预防提供了新的干预靶点。
(7)未来研究方向
1.利用转基因小鼠模型和类器官培养系统验证多组分配方的有效性
2.开发基于血液和脑脊液生物标志物的早期诊断方法
3.探索环境干预与营养干预相结合的预防策略
4.研究线粒体功能障碍与神经元退化的关联机制
5.开发靶向神经炎症的精准调节方案
2.儿童发育障碍
儿童发育障碍如自闭症谱系障碍、注意力缺陷多动障碍、抽动障碍等,严重影响儿童的认知、社交和行为发展,给家庭和社会带来巨大负担。这些疾病的病因复杂,涉及遗传因素、环境因素和营养因素的多层次互作。研究团队采用系统生物学方法,深入探索这些障碍的分子机制和潜在干预策略。
(1)多维数据整合分析
研究团队构建了一个涵盖神经发育全周期的多维数据分析平台,整合了发育神经生物学、表观遗传学、营养组学和环境毒理学等多学科数据。通过对468条神经发育相关通路、273条环境响应通路和185条营养代谢通路的系统分析,绘制了儿童发育障碍的分子网络图谱。这一分析揭示了神经发育过程中的关键时间窗口和敏感节点,特别是胚胎期神经管形成、胎儿期神经元迁移、出生后突触修剪和髓鞘形成等关键阶段。研究表明,这些时期的分子调控网络异常与发育障碍的发生密切相关。
(2)基因-环境-营养互作网络
通过整合分析,研究团队鉴定出523个与儿童发育障碍相关的基因,并将其分类为核心致病基因、易感基因和修饰基因三个层次。这些基因主要富集在神经元分化、突触形成、轴突导向、神经递质代谢和胶质细胞功能等生物学过程。研究发现约78%的相关基因存在环境因素响应元件或营养敏感调控区域,表明基因-环境-营养的三维互作在疾病发生中发挥关键作用。团队构建的互作网络模型显示,某些环境毒素(如重金属、内分泌干扰物)和营养因素(如叶酸、维生素D、ω-3脂肪酸)可通过表观遗传修饰机制影响关键基因的表达,进而干扰神经发育进程。
(3)表观遗传调控机制
研究团队对发育障碍患儿的外周血和动物模型的脑组织进行了表观遗传组学分析,发现DNA甲基化、组蛋白修饰和非编码RNA调控在疾病发生中扮演重要角色。特别是,某些环境因素可通过改变特定基因的甲基化状态,影响神经元分化和突触可塑性。为理解环境因素如何"编程"神经发育提供了分子机制,也为早期干预策略的开发指明了方向。团队发现,某些营养因子(如叶酸、胆碱和DHA)可通过逆转特定的表观遗传改变,潜在地缓解发育异常。
(4)肠-脑轴与微生物组研究
基于近年来微生物组学研究的突破,团队特别关注了肠-脑轴在儿童发育障碍中的作用。通过整合分析肠道微生物组数据和代谢组学数据,研究人员发现发育障碍儿童的肠道菌群多样性降低,短链脂肪酸产生减少,肠道屏障功能受损。这些改变可能通过多种途径影响神经发育,包括神经炎症反应、神经递质代谢、迷走神经信号传导和微生物代谢产物直接作用等。研究表明某些天然活性成分可通过调节肠道微生物组平衡,间接改善神经发育和行为表现。
(5)多靶点干预策略
基于上述系统分析,研究团队筛选了上百种潜在的天然干预物质,包括多种植物多酚、ω-3脂肪酸、益生菌、益生元和常量/微量营养素等。通过网络药理学分析,团队评估了这些物质对发育障碍相关通路的调节潜力和网络干预效率。体外实验数据显示,某些组合配方可能通过多重机制发挥协同作用,包括减轻氧化应激、调节神经炎症、促进神经营养因子表达、改善线粒体功能和恢复突触可塑性等。特别是,一些植物源多酚类化合物和特定微量营养素的组合显示出良好的神经保护作用和行为调节潜力。
(6)早期诊断与个体化干预
研究团队还致力于开发基于多组学数据的早期诊断标志物系统。通过整合基因组、表观基因组、代谢组和微生物组数据,正在构建一个多层次的生物标志物网络,旨在实现发育障碍的早期识别和亚型分类。这一多维诊断系统有望支持个体化干预策略的制定,使干预措施能够针对不同亚型的分子病理特征进行精准调整。研究表明,早期干预对发育障碍的预后至关重要,因为神经系统的可塑性在生命早期最为显著。
(7)未来研究方向
1.开展前瞻性队列研究验证关键生物标志物的预测价值
2.利用神经发育类器官模型评估多组分配方的神经发育调节作用
3.探索基于肠-脑轴的干预策略与传统行为干预的协同效应
4.研究关键发育窗口期的精准营养干预方案
5.开发适合不同发育障碍亚型的个体化干预策略
6.建立家庭-学校-医疗机构联动的综合干预体系
3.自身免疫性疾病
自身免疫性疾病如类风湿关节炎、系统性红斑狼疮和过敏性疾病等,是免疫系统失调导致的慢性疾病。现有治疗方法往往伴随着严重的副作用,且难以实现长期缓解。麦基洗德生物数据系统构建了免疫系统的动态平衡模型,试图找出能够精准调节免疫平衡的天然活性成分。
(1)临床数据驱动的研究方向
麦基洗德团队在过敏领域拥有丰富的临床实践经验,特别是自主研发的"利敏舒"益生菌产品已在临床应用中取得显著成效。生物数据系统整合了来自全国多中心的基层医疗机构的数千例患者的临床数据,包括消化系统过敏(如牛奶蛋白过敏)、皮肤过敏(如湿疹、荨麻疹)、呼吸系统过敏(如过敏性鼻炎、哮喘)等多种表现形式。系统分析显示,约65%的过敏患者同时存在多系统症状,这一发现促使团队将过敏性疾病视为全身性免疫调节紊乱,而非局部问题。通过生物数据系统的整合分析,团队识别出了不同过敏表型的分子特征和潜在干预靶点。
(2)微生物组-免疫轴的数据整合
研发团队特别关注微生物组与免疫功能的关联数据。通过整合临床样本的微生物组测序数据、免疫指标和临床效果,系统构建了一个微生物组-免疫互作网络模型。这一模型揭示了特定菌群组合与免疫平衡之间的关系,为开发精准的微生态免疫调节策略提供了理论基础。数据分析显示,利敏舒中的特定菌株组合能够通过调节肠道菌群平衡,影响多种免疫参数,包括T辅助细胞亚群比例、调节性T细胞数量、炎症因子水平和分泌型IgA产生等。这些发现被系统记录并用于指导产品优化和个性化应用方案设计。
(3)天然活性成分的免疫调节数据库
作为生物数据系统的核心组成部分,团队构建了一个包含超过500种天然活性成分免疫调节作用的专业数据库。通过整合文献数据、体外实验结果和公司商业化产品利敏舒的临床观察,系统对这些成分的免疫调节机制、有效剂量和安全性进行了系统评估,识别出了一系列具有调节T细胞亚群平衡、抑制过度炎症反应潜力的植物源活性成分。通过网络药理学分析,系统预测了这些成分与免疫相关靶点的相互作用,并根据作用模式进行了分类,为配方优化提供了数据支持。
(4)临床验证数据的反馈循环
生物数据系统不仅整合已有知识,还建立了临床验证数据的反馈机制。团队系统收集了利敏舒在不同过敏表现中的历年临床效果数据,包括:婴幼儿牛奶蛋白过敏所致慢性腹泻的治疗研究,总有效率达92.5%;婴儿慢性湿疹的联合治疗研究,总有效率达95.35%;特应性皮炎的辅助治疗研究,疾病复发率从42.86%降至21.43%;儿童变应性咳嗽的联合治疗研究,总有效率达93.67%;常年性过敏性鼻炎的治疗研究,症状评分显著降低。这些临床数据被系统化记录并反馈到生物数据系统,用于验证预测模型的准确性,并指导下一代产品的研发方向。
(5)基于数据驱动的产品升级计划
依托生物数据系统的分析结果,麦基洗德团队计划在2025年底完成"利敏舒"产品的升级和配方优化。原来针对所有过敏症状的单一产品将演进为针对特定过敏表现的精准系列产品,使干预更为精准和具体。生物数据系统通过分析不同过敏亚型的分子特征和临床表现,为每种配方的设计提供了数据支持。初步的临床观察表明,这种数据驱动的精准化策略能够进一步提高临床效果,缩短症状改善时间。
(6)未来研究与系统升级
1.扩展数据采集范围,纳入更多自身免疫性疾病类型和患者亚群
2.开发针对不同过敏表现的精准配方系列
3.研究肠道菌群代谢产物与免疫调节的分子机制
4.探索过敏-微生物-营养三维互作网络
5.建立过敏性疾病早期预警和干预体系
6.开发适合中国人群特点的过敏风险评估工具
7.研究心理因素对免疫功能的影响及其调节策略
开放合作,共建生物医药创新生态
麦基洗德坚信,解决人类健康挑战需要全球科研力量的协作。公司计划在2027年数据系统彻底建成后,向全球科研机构开放数据资源和分析工具,促进生物医药领域的开放创新。这不是简单的数据共享,而是建立一个互惠互利的创新生态系统。公司将提供标准化的API接口,允许全球高校或科研机构免费、安全地访问特定数据集和分析工具,合作伙伴可以基于这一平台开展自己的研究项目,同时贡献新的数据和见解,形成良性循环。"我们希望这个系统能够成为连接全球科研力量的桥梁,让更多研究者能够基于这一平台开展创新研究,共同推动生物医药科学的发展。"李天琦表示,"麦基洗德愿意成为这一生态系统的建设者和参与者,与全球合作伙伴一起,为人类健康事业贡献力量。"
1.未来三年目标和计划
2025年(第一阶段进行中)
完成初步生物多样性数据库建设,收录3万种生物样本;整合70万种具备药用价值的生物活性成分数据;完成百万级科研文献的结构化处理与知识提取;启动1~2家科研机构的试点合作项目(上述提及的量化指标,在第一阶段仅作为系统测试、功能验证和算力测试的基础数据集和初试样本)。
2026年(第二阶段)
大幅扩充生物多样性数据库,目标收录>15万种生物样本;扩展生物活性成分数据库至300万种,重点关注多源交叉验证;整合500万级科研文献数据,实现跨语言、跨学科的知识挖掘;完成临床数据与基础研究数据的深度整合;开发并优化疾病特异性的预测模型;在神经退行性疾病领域完成2-3个候选配方的动物实验验证;在儿童发育障碍领域建立完整的早期筛查与干预体系;在自身免疫性疾病领域推出升级版"利敏舒"相关的系列产品;扩大合作网络至3~5家国内外研究机构。
2027年(系统全面建成)
实现生物多样性数据库的全面覆盖,目标收录69万种以上生物样本;生物活性成分数据库扩充至1000万种以上,建立全球最全面的天然活性成分数据库;完成2000万级科研文献的结构化处理与知识图谱构建;完成全部数据系统架构建设与功能优化;实现AI辅助的活性成分筛选与配方设计全流程;向全球科研机构开放标准化API接口;在三大疾病领域各产出1-2个具有临床转化潜力的研究成果;启动至少一个多中心临床研究项目;建立完整的知识产权保护与成果共享机制;形成10家以上研究机构参与的全球合作网络。
2.三大疾病领域目标
神经退行性疾病领域
2025年完成多组分配方的体外筛选与初步机制研究;2026年完成转基因小鼠模型的有效性验证与安全性评估;2027年确定1~2个具有临床转化潜力的候选配方,并启动早期人体试验准备工作。
儿童发育障碍领域
2025年建立完整的生物标志物评估体系与早期筛查工具;2026年完成基于肠-脑轴的干预方案在类器官模型中的验证;2027年形成一套综合干预方案,并在3-5家医疗机构开展临床观察研究
自身免疫性疾病领域
2025年完成针对不同过敏表现的精准配方开发与初步临床验证;2026年推出升级版"利敏舒"系列产品,覆盖多种过敏亚型;2027年建立完整的过敏风险评估与精准干预体系,并在全国多中心开展应用研究
通过这一系列目标明确、时间节点清晰的计划,麦基洗德将逐步实现从数据到知识、从知识到产品、从产品到健康的转化路径。我们的使命是深入理解罕见病的分子机制,为这些长期被忽视的疾病群体提供更有效的干预方案。值得强调的是,第一阶段的数据收集仅是系统功能验证的基础,真正的大规模数据整合将在第二和第三阶段实现。这体现了麦基洗德"先建立框架、验证功能,再逐步扩充数据"的务实发展策略,确保系统架构的稳健性和可扩展性,为后续海量数据的高效处理和深度挖掘奠定坚实基础。
正如业界前辈屠呦呦所言:"青蒿素是中医药献给世界的一份礼物。"秉承这一传承创新精神,麦基洗德致力于通过整合全球生物多样性资源、多组学数据和临床实践经验,探索天然活性成分的治疗潜力。我们坚信,科学的价值不在于追逐商业利益,而在于解决人类面临的健康挑战。通过系统性地连接自然界的生物智慧与现代医学科技,我们期望为罕见病患者带来新的希望,为生物医药创新贡献中国智慧和中国方案。
关于麦基洗德
烟台麦基洗德生物工程有限公司成立于2017年,总部位于山东龙口,是一家由90后为主的年轻团队组成的企业,公司成员来自世界各地,专业背景涵盖数学、计算机科学、生物信息学、细胞与分子生物学、药理学、药剂学、病理学、营养学、临床医学、生物技术、环境科学、视觉艺术、信息艺术和传统美术等多个学科。
作为一个新兴的创业团队,麦基洗德以独特的视角和方法探索生物多样性及天然活性成分的研究。公司正致力于构建计算生物学、天然产物化学和药效评估等研发平台,逐步形成从数据挖掘、活性成分发现到功能验证的完整研究路径。公司重视技术创新,科研经费平均占企业利润比80%以上,并积极寻求与高校、研究机构及医疗机构的合作机会,希望通过开放协作的方式,为生物医药领域带来创新思路。
纵使前路漫漫,我们依然投入全心全灵、不遗余力、不计代价,只为解锁更多疾病的神秘密码。在商业利益与科学真理的抉择中,在短期回报与长远价值的权衡间,在个体荣誉与集体福祉的天平上,我们始终选择真理、长远与众生。因为我们坚信,自然界的每一种生物、每一个分子,都可能是改变人类命运的关键,当青蒿素还沉睡在古老医书的字里行间,当青霉素尚未从那片意外的霉菌中被发现,世界也曾对这些改变人类命运的物质一无所知,然而,正是那些最初难以理解的探索,往往孕育着最伟大的突破。
相关阅读
-
纠结两岁宝宝喝什么奶粉好?先搞懂这些再选不踩坑
两岁宝宝喝什么奶粉好?两岁时一般已经过渡到三段奶粉了,但很多家长依然疑惑三段奶粉怎么选、三段什么奶粉好等问题。其实选奶不能盲目跟风,无论是宝宝三段奶粉怎么选择或宝宝三段喝什么奶粉好,核心都要围绕宝宝的 ...11-27 -
麦施美学旗下重要星品XEOMIN®思奥美®和RADIESSE 芮得怡上市会正式召开
重塑美学体验引领技术创新全球最大的专注医疗美容的企业MerzAesthetics®麦施美学(以下简称麦施美学)宣布旗下两款重要新品XEOMIN®思奥美®和RADIESSE芮得怡召开上市发布会,这标志着麦施美学在中国市场面部注射类 ...11-25 -
全省十佳!惠州口腔医院荣获“年度十大健康新媒体”
近日,第六届南方健康传播与创新大会传来捷报,惠州口腔医院公众号,凭借在健康传播领域的突出表现.成功斩获“年度十大健康新媒体(专科医院类)”。这场由广东省医学会主办,广东省医学会健康传播自媒体联盟、广东 ...11-25 -
断奶后喝什么奶粉好?这几款满足宝宝成长的多维需求
断奶是宝宝成长中的重要转折,而此时家长最关心的便是断奶后喝什么奶粉好。断奶后,奶粉成为宝宝的重要营养来源之一,想要满足生长发育需求,营养成分自然是越全面均衡越好。若奶粉营养单一或配比不当,易导致宝宝营 ...11-25 -
婴儿便秘换什么奶粉比较好?先辨因再选奶
宝宝便秘是家长育儿路上的高频难题,宝宝排便时小脸憋红、哭闹不止,便便干结如羊粪球,不仅让宝宝痛苦,也让家长焦虑不已。不少家长认为,宝宝便秘是由奶粉引起,毕竟奶粉作为宝宝的关键营养来源之一,其配方容易影 ...11-25 -
国产OK镜品牌怎么选择,个性化定制成竞争焦点
近年来,随着青少年近视防控需求日益迫切及个性化医疗理念的普及,角膜塑形镜(OK镜)已从单一的视力矫正工具,逐步发展成为集个性化定制、近视防控管理于一体的视光解决方案。2025年的OK镜市场预计将持续扩大,但品 ...11-24 -
卸下疼痛包袱:花生医疗吴美艳主任连答「无痛分娩14问」
2017年,陕西榆林,一位待产孕妇从医院五楼一跃而下,带着未出世的孩子,永远离开了世界, 她的绝望,是对产痛无人理解的无声控诉。“忍一忍就过去了”——这句看似安慰的话语,实则折射出千百年来女性在生育中被迫 ...11-24 -
牙黄怎么办?2025值得关注的美白牙膏红榜研选
牙齿泛黄是普遍存在的口腔健康问题。要有效应对,需从其科学成因入手。口腔医学研究表明,咖啡、茶、酱油等饮食中的色素,并非简单附着。它们会与口腔细菌分泌的黏性物质(牙菌斑生物膜)结合,形成稳固的复合物,最 ...11-24 -
重庆送子鸟医院曝光!口碑见证实力,用专业守护“孕”望
在重庆的医疗版图中,有这样一家医院,它不靠炒作博眼球,只用疗效赢口碑;它没有负面曝光的纷扰,却有无数家庭重拾欢笑的故事。它就是重庆送子鸟医院,专注生殖健康领域多年,以专业医术、暖心服务,成为众多不孕不 ...11-24 -
儿童咳嗽就吃止咳药?这些用药常识家长必须懂
入冬后,呼吸道疾病进入高发期,抵抗力较弱的孩子更是易感人群。“孩子流鼻涕咳嗽怎么缓解”“咳嗽有痰要吃药吗”“会不会咳成肺炎”—— 妈妈群里,类似的疑问频频出现,不少家长面对孩子咳嗽便手足无措。其实,家 ...11-22